Métodos clásicos de secuenciación
MÉTODO ENZIMÁTICO DE SANGER
Se diseña un oligonucleótido sintético de unos 17-20 bases complementario de la cadena de ADN que se quiere secuenciar y situado a unos 20-30 bases de distancia del comienzo de la secuencia que se quiere leer. Si este oligo se diseña fuera de la región de clonaje de los plásmidos podrá emplearse el mismo oligo para secuenciar distintos insertos.
El dúplex formado entre el oligo y el ADN complementario de cadena sencilla se convierte en sustrato de la ADN polimerasa I que va a extender la cadena desde grupo OH libre del extremo 3´ del oligo, incorporando dNTPs y copiando el molde de ADN al sintetizar la cadena complementaria. Como enzima se emplea el fragmento Klenow de la ADN polimerasa I que carece de actividad exonucleasa 5´- 3. También pueden emplearse otras polimerasas como la Taq polimerasa o la polimerasa del fago T7.
Durante la secuenciación se llevan a cabo cuatro reacciones de síntesis separadas incluyendo en cada una de ellas pequeñas cantidades de los dideoxinucleótidos (ddNTP: ddGTP; ddATP, ddCTP, ddTTP) que carecen de extremo 3´ OH libre y que al incorporarse en la cadena de ADN que se esta sintetizando acaban con la elongación de la misma.
La incorporación al azar de un ddNTP en competición con el dNTP correspondiente, implica la formación de una mezcla de cadenas de distintas longitudes, todas ellas empezando en el extremo 5´ y acabando en todas las diferentes posiciones posibles donde un ddNTP puede incorporarse en lugar de un dNTP. El promedio de longitud de las cadenas puede alterarse modificando la relación dNTP/ddNTP en la mezcla de reacción. Por ejemplo, aumentando la concentración de ddNTP aumenta el número de cadenas de pequeña longitud al aumentar la frecuencia de incorporación de este tipo de nucleótidos.
La sustitución de uno de los dNTPs por el mismo nucleótido marcado radiactivamente permite la visualización de las bandas de distinta longitud en un gel de poliacrilamida donde cada una de las reacciones se carga en un carril. En el gel la separación de las distintas bandas se produce en función de su tamaño y la secuencia puede determinarse leyendo las bandas de los cuatro carriles. En este tipo de geles pueden leerse hasta 300 bases. En carreras más largas la lectura puede llegar a ser de hasta 500 bases.
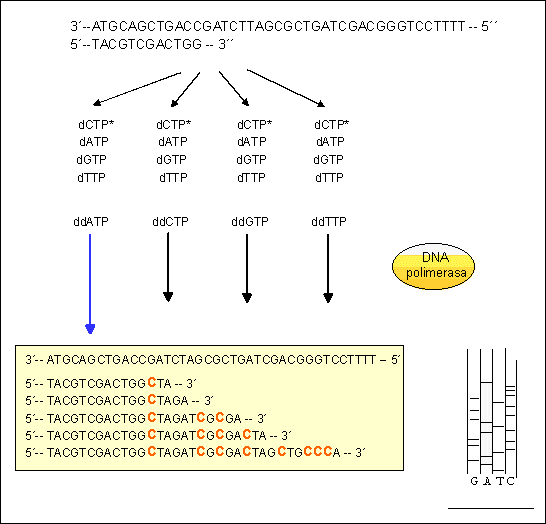
| Método de secuenciación de Sanger |
IIB - CSIC