Otros métodos de secuenciación automática
SECUENCIACION DE ADN EMPLEANDO MICROARRAYS
Principio:
Existen muchas variedades de microarrays o ADN-chips como también se les denomina. Desde los microarrays "artesanales" realizados en el laboratorio a los ofrecidos por compañías de biotecnología, el principio que rige su diseño es el mismo: la complementariedad de las cadenas aisladas de ADN: o propiedad de las citadas cadenas de hibridarse con otras cadenas aisladas de ADN que posean una estructura complementaria.
Básicamente un microarray consiste en una matriz de "pocillos" microminiaturizados sobre un substrato de vidrio en donde se implantan, utilizando diversas técnicas, cadenas simples de oligonucleótidos. El poder adherir una cadena corta de oligo sobre una superficie plana es decisivo en el diseño de los microarrays.
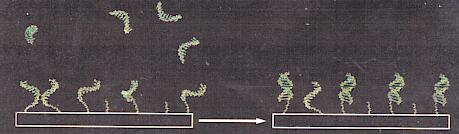
(Molecular interactions on microarrays, Edwin Southern..., Nature Genetics, january 1999, pp..5)
En la figura se muestran esquemáticamente unas pocas celdas de un microarray. En cada celda, adherida por uno de sus extremos, existe una determinada cadena de oligonucleótido. (zona inferior izquierda de la imagen) Si se pone el microarray en contacto con una mezcla de cadenas simples de ADN a identificar, marcadas fluorescentemente, (zona superior izquierda) éstas se hibridarán con su oligo complementario. (zona derecha).
Dado que conocemos la secuencia y distribución de los oligonucleótidos en el microarray podemos, al excitar el microarrray con un laser y estar las cadenas hibridadas fluorescentemente, conocer la ubicación de las cadenas de las mismas y su secuencia..
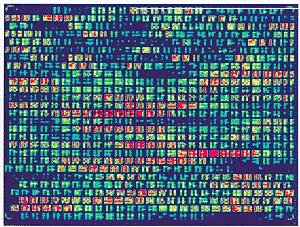
Visualización típica de una secuenciación mediante microarrays
Fabricación;
En la fabricación de microarrays se emplean técnicas fotolitográficas análogas a las utilizadas en microelectrónica. Un sustrato de vidrio se trata químicamente con determinados grupos reactivos para permitir la implantación de los oligonucleótidos sobre el mismo; a continuación se deposita sobre el substrato una película fotodegradable y mediante la utilización de una plantilla y un haz luminoso, se crea la estructuura de celdas del microarray.
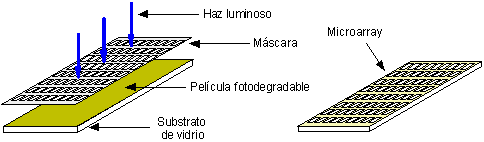
Fabricación de las celdas del microarray
- Implantar en cada celda, mediante un brazo robotizado, el oligonucleótido presintetizado que corresponda.
- Sintetizar en las propias celdas, mediante ciclos sucesivos de síntesis, los oligonucleótidos correspondientes.
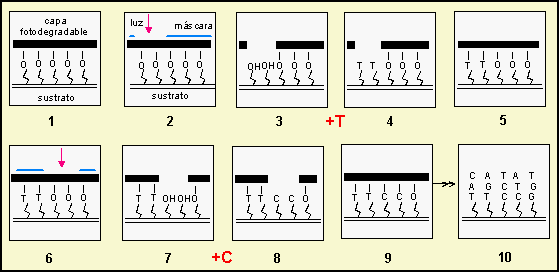
El proceso de síntesis "in situ" de los nucleótidos en el microarray se realiza en las siguientes fases:
- Elaboración previa de un mapa de distribución del tipo de oligonucleótido correspondiente a cada celda del microarray.
- Preparación del sustrato y deposición de una película fotodegradable. (1)
- Aplicación de una máscara que permita eliminar selectivamente la película protectora en las zonas del microarray correspondiente a un determinado nucleótido. (2 y 3)
- Incubación química y acoplamiento del nucleótido previsto (4).
- Una nueva capa fotodegradable es aplicada sobre el microarray (5)
- Se repiten los pasos anteriores para cada nucleótido hasta obtener la secuencia prevista
- Se elimina definitivamente la película fotodegradable (10).
Ejemplo de utilización:
La localización e identificación de diferencias de secuencia en relación con diferencias fenotípicas constituye la base de distinción entre estado normal y enfermedad. Dada una secuencia determinada de una región de ADN es posible diseñar cuatro sondas diferentes por cada posición. Una de ellas será perfectamente complementaria de la secuencia de referencia mientras que las demás lo serán en todas las posiciones salvo en la de la posición que se interroga donde esa base ha sido sustituida por las otras tres posibles.
Tras la incubación con la secuencia de referencia la mayor señal de hibridación fluorescente corresponderá a la secuencia 100% complementaria. Si la muestra presenta alguna mutación en la posición interrogada la mayor señal corresponde a la hibridación con la correspondiente secuencia complementaria. Mil sets de cuatro sondas cada uno se emplean para interrogar las mil posiciones de la secuencia. Esta estrategia funciona especialmente bien para la detección de polimorfismos en el ADN indicando tanto la posición de la sustitución como la naturaleza de la misma.
IIB - CSIC