Curso introduccion al Linux. |
|||
|---|---|---|---|
Linux es un sistema operativo. Los sistemas operativos sirven para interaccionar con el usuario del ordenador y poder realizar tareas como programar, jugar, ejecutar programas y utilidades, encender y apagar el ordenador, leer cd-roms, producir sonido a partir de información digital.
Es decir, el sistema operativo gestiona los recursos de la computadora, esto es: Medios de almacenamiento (discos duros, cd-roms, disquetes, ZIPs...), la memoria, la pantalla gráfica, en fin, todo. Sin un sistema operativo, una computadora no es interactiva y sería inutilizable salvo como super calculadora. Además sería imposible que un usuario no especializado manejara la computadora.
Linux es una implementación de los Unix comerciales desde cero, realizada por el finlandés Linus Torvalds cuando era un estudiante. Desde entonces ha evolucionado mucho, gracias al esfuerzo de la comunidad de internautas que se ha implicado en el proyecto de su constante mejora y evolución. La mentalidad que ha permitido este fenómeno se denomina proyecto GNU, cuya filosofía, apoyada por la FSF (Free Software Foundation) desencadenó entre muchas otras cosas, la aparición de este sistema operativo gratuito, de libre distribución y de potencia igual o superior a las soluciones de las grandes marcas comerciales.
Actualmente Linux es una versión más de Unix, quizá la más diferenciada de todas y se puede utilzar para cualquier entorno, ya sea el de grandes servidores de proceso de datos, de servicios de red e internet o como workstation. En este último caso, quizá es un sistema que requiere algo más de esfuerzo por parte del usuario medio que otros sistemas como Windows, pero hay que decir en su favor que en Linux los programas no se cuelgan ni el sistema se bloquea y que cuando algo se sabe hacer, se sabe hacer bien y comprendiendose desde el principio hasta el final lo que se esta haciendo. Como siempre la computadora puede fallar, pero el sistema operativo es quizá a fecha de hoy, el más estable conocido.
Como estación de trabajo, Linux es muy apreciado en los entornos técnicos, ingenieriles y sobre todo científicos en general.
En entornos de servidores de proceso de datos, la razón fundamental es el alto rendimiento de Linux como sistema operativo paralelizado. Esto puede ser por medio de la instalación del sistema en computadoras con dos o más microprocesadores. Pero también resulta muy interesante implementar Clusters de ordenadores con Linux, solución muy barata para tener potencia semejante al de las computadoras multiprocesador. La diferencia es, una computadora que tiene 16 microprocesadores dentro, o 16 ordenadores normales instalados para que procesen datos como uno solo de 16 micros. La segunda opción es siempre menos eficiente, pero más barata y siempre se le pueden ir añadiendo más y más equipos en principio sin límite.
En entornos de servicios de red, Linux es un sistema que maneja el stack TCP/IP de manera envidiable y de forma nativa, pues toda La Internet y el protocolo TCP/IP de red se desarrollo sobre sistemas operativos Unix, y Linux es una implementación de Unix.
Para desarrollo, Linux ofrece un sistema con multitud de utilidades, compiladores de todos los lenguajes de programación, y una transparencia en su código de categoría académica. La diferencia entre trabajar en desarrollo con Linux o hacerlo con Windows sería la misma que encontraríamos en otro contexto entre investigar para una universidad o para una empresa privada.
Como estación de trabajo para investigadores científicos, es muy útil pues ofrece un sistema potente de cálculo, proceso de datos, de programación de aplicaciones propias en cualquier lenguaje (lo que significa que puedes ejecutar y compilar las aplicaciones desarrolladas por otros laboratorios) y mucho más. El único problema es que como todo lo bueno, hay que aprender a utilzarlo y Linux es extenso, muy extenso. Pero no falta la documentación incorporada en la propia instalación del sistema. ¿Que se puede parecer más a un laboratorio de investigación? Además con Linux, es posible hacerlo todo, con otros sistemas operativos no. Si algo se puede hacer en el mundo de la informática, alguien lo ha hecho en linux o lo está haciendo en estos momentos para ponerlo al alcance de la comunidad virtual.
EJEMPLO: El util comando man
Existen varias distribuciones de Linux más o menos conocidas, aunque cualquiera podría crear su propia distribución. Las más conocidas son Debian, Red Hat y Suse. No comentaremos otras distribuciones por considerarlas menores y/o obsoletas. De estas tres, cabe reseñar que Red Hat es la más compatible, suficientemente innovadora y sencilla de instalar para el usuario medio. Suse suele ser algo más cómoda para el usuario, pero menos extendida que Red Hat y finalmente Debian es el claro exponente del verdadero GNU/Linux, es decir solo hay un verdadero Linux en todos los sentidos tanto técnicos como filosóficos y ese es Debian. Es la distribución más estable, la que más software libre y no libre tiene a su disposición y la que mejores características técnicas ofrece. Pero en su contra esta que su diseño se centra sobre todo en lo que los informáticos consideran "bueno", que por desgracia para los usuarios no avanzados, no sele ser lo mismo que ellos aprecian.
En el IIB tenemos 2 servidores para el correo electrónico, las bases de datos de personal, las cuentas de usuario, el servidor web, un mirror de tucows, el DNS... En fin, todo lo que se necesita para trabajar en una red interna y en internet. Estos servidores Linux tienen instalado un sistema Red Hat.
Adicionalmente va a instalarse un servidor dedicado a bioinformática y biocomputación, que muy probablemente tendrá instalado un sistema Debian, puesto que es la distribución mas estable y que más aplicaciones ofrece a la comunidad científica.
Los servidores Linux permiten el acceso por red de los usuarios dados de alta en el IIB. El nombre de usuario y la clave que se necesitan son las mismas que usamos para entrar en el correo electrónico.
Es el método tradicional de conexión a un terminal Unix. Este comando esta presente en todos los sistemas operativos. Tiene un problema para usarse sin cuidado: Dado que el protocolo TCP/IP suele ir sobre redes Ethernet, de tipo público, al no ser una conexión encriptada, todo el mundo puede ver lo que tecleamos si se preocupa de capturar los paquetes públicos TCP/IP que se pueden ver desde su tarjeta de red (la que usa para escuchar y conectarse a la red que esta en cada ordenador).
El ssh consiste, por decirlo de alguna manera, en un telnet protegido por encriptación. Esto quiere decir que la información que se envia por la red desde nuestro ordenador al servidor necesaria para conectarse ambos, viaja por los cables de red de un lado a otro en formato encriptado, usando una clave muy dificil de desproteger por medio de hackers o agentes espías.
Por lo demas, es al igual que el telnet, se necesita un nombre de usuario y una calver para acceder.
El programa que utilizamos en Bioinformática es el Putty para PC, y el MacSSH para Mac, pero existen muchos otros para todos los gustos (salvo quizá en el caso de Mac).
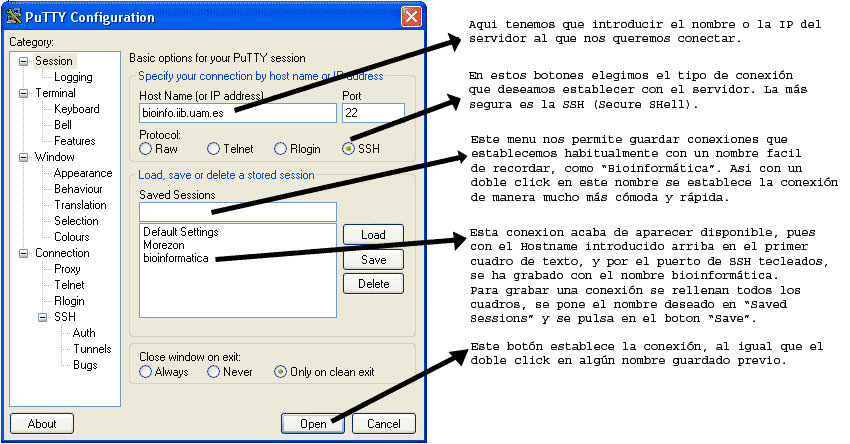
Aqui podemos ver como se conecta un usuario al servidor usando este programa paso por paso:
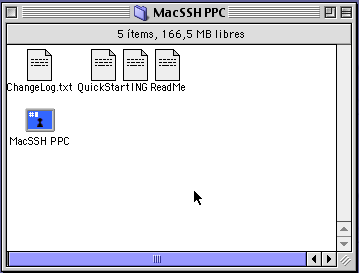
1 -. Seleccionamos con el raton la aplicación MacSSH:
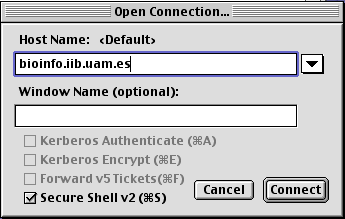
2 -. Con el menu elegimos la opción "Open Connection" y nos aparece esta ventana:
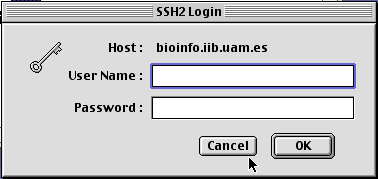
3 -. En ese momento debemos autentificarnos contra el login del servidor con nuestro nombre de usuario y la contraseña habitual:
4 -. Finalmente entramos en la shell del sistema y podemos empezar a trabajar con el servidor.
Es importante conocer las características de tu cuenta de usuario, dado que es el corazón de toda actividad e interacción que tú, como parte del personal del IIB, puedes tener con los servicios de informática del centro (correo, web, internet, intranet, acceso al servidor de bioinformática, cyberlar...).
Mucha gente obvia la existencia de estas cuentas en los servidores, otras personas incluso lo desconocen. La intención de este apartado es destacar la importancia de lo que es quizá la característica más potente de todo el servicio que ofrecemos: Poseer una cuenta de usuario en un servidor Linux.
Esto nos permite trabajar con el servidor en tareas como el calculo científico, análisis de secuencias, datos de microarrays, determinación de estructura de proteinas, filogenia.... desde un entorno de trabajo tipoUnix, que es sumamente cómodo y potente para todo especialista en materias de alta necesidad computacional, como son los científicos: Personas que buscan el dominio de la materia y la total comprensión de lo que hacen, asi como la posibilidad de convertir sus ideas en resultados empíricos a través de la lógica y el método y no siendo dependientes de soluciones comerciales "oscuras".
Luego la cuenta de usuario nos permite:
1- Utilizar todos los servicios de la red informática del Instituto.
2- Ejecutar programas (y programarlos si queremos) para manipular, obtener y descodificar datos importantes en experimentación biomédica (esto es básicamente la bioinformática y la biocomputación).
Manipulando datos podemos obtener interesantes cálculos, ya sean estructurales, ya sean en rutas metabólicas, en enzimología...
Obteniendo datos, se pueden contrastar hipótesis, crear otras nuevas, planificar un método o el abordaje de un tema para su fase experimental con criterios en la mano. O incluso a nivel teórico, comprobar esas hipótesis.
Descodificando datos, como pueden ser los obtenidos de arrays o directamente el genoma, se puede comprender el lenguaje último de la de la biología, de la vida. Quizá en éste punto estemos contemplando el que será en los proximos años terreno de juego para la mayoría de los proyectos científicos en todo el mundo. Encontrar todos los genes y saber como funcionan.
Desde "Entorno de Red" con el explorador de archivos, seleccionamos lo que puede verse abajo:
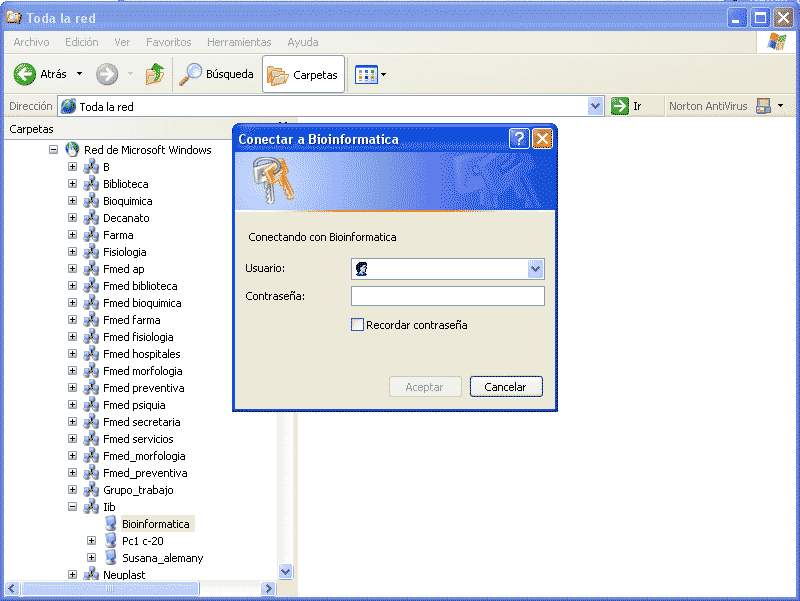
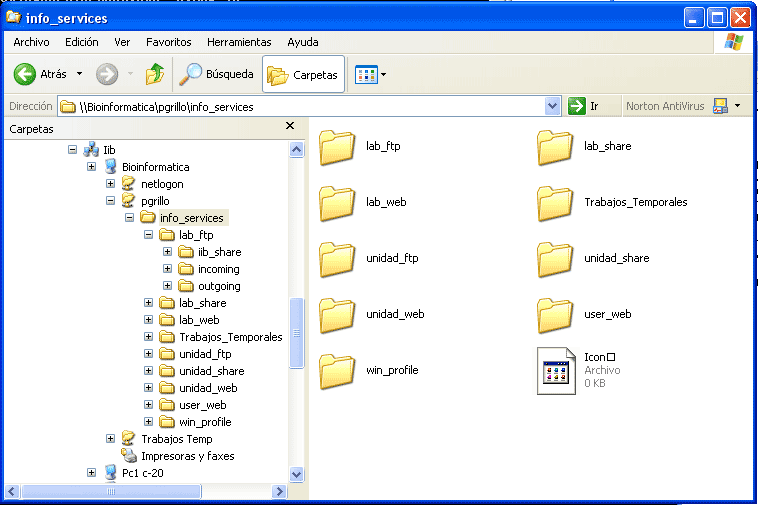
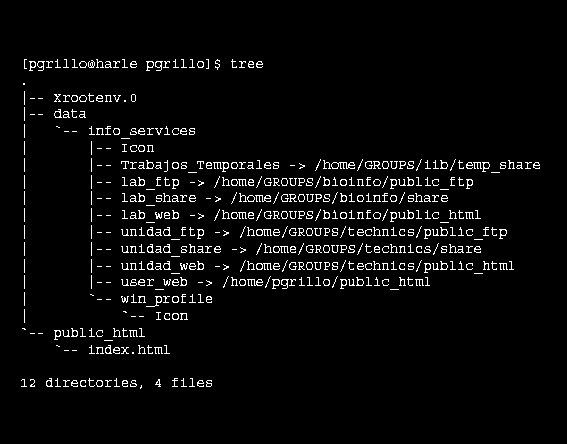
En Linux, a traves de SSH o Telnet, podemos ver la figura de la derecha con fondo negro, utilizando el comando tree, que muestra el arbol de directorios desde la raiz que se le de como parámetro o desde el lugar donde nos encontremos en el momento de su ejecución. A la izquierda vemos lo mismo, pero desde windows a traves del servidor de ficheros:
Lo importante es saber que ambas cosas, son los mismos ficheros y directorios, accedidos de maneras diferentes ¡Y mostrados de formas muy diferentes!
 |
 |
Para entrar y salir de directorios se utiliza el comando cd con el argumento del directorio o ruta a la que queremos acceder o entrar, o con ".." si queremos saltar al directorio anterior al que ocupemos en el momento de la ejecucion de cd.
Ej:
[pgrillo@bioinfo pgrillo]$ cd data
O lo que es lo mismo:
[pgrillo@bioinfo pgrillo]$ cd /home/cquijano/data
para volver atrás:
[pgrillo@bioinfo pgrillo]$ cd ..
Es exactamente lo mismo que hemos hecho antes con el comando cd. Hay que tener en cuenta ciertas cosas cuando se ejecutan programas en la linea de comandos:
La forma básica de ejecutar una orden consiste en teclear su nombre y pulsar "entrar". Pero lo más común es que todas las ordenes permitan opciones que modifican su ejecución. Estas opciones comienzan por el caracter - (menos) la mayoría de las veces. A veces es necesario que los parámetros esten separados por espacios para ser reconocidos.
Además, en cualquier sistema tipo UNIX tenemos que tener mucho cuidado con las letras mayúsculas y minúsculas, pues al contrario que otros sistemas operativos, todo UNIX las distingue.
Ejemplo de ejecución de orden con parámetros modificadores:
[pgrillo@harle pgrillo]$ ls
-l
total 3
-rw-rw-r-- 1 pgrillo pgrillo 721 Mar 10 2000 Xrootenv.0
drwxr-x--- 5 pgrillo pgrillo 1024 Mar 24 2000 data/
drwxr-xr-x 2 pgrillo pgrillo 1024 Sep 2 1999 public_html/
[pgrillo@harle pgrillo]$ ls -l data
total 1
drwxr-x--- 4 pgrillo pgrillo 1024 Jun 7 2002 info_services/
[pgrillo@harle pgrillo]$ ls -l Xrootenv.0
-rw-rw-r-- 1 pgrillo pgrillo 721 Mar 10 2000 Xrootenv.0
[pgrillo@harle pgrillo]$ ls -li Xrootenv.0
1046626 -rw-rw-r-- 1 pgrillo pgrillo 721 Mar 10 2000 Xrootenv.0
[pgrillo@harle pgrillo]$ LS -L
bash: LS: command not found
[pgrillo@harle pgrillo]$
Toda la documentación y ayuda acerca de EMBOSS esta en su web
oficial.
En el IIB tenemos la siguiente forma de activar las variables de entorno de
Emboss necesarias para poder ejecutarlo:
[pgrillo@harle pgrillo]$ emboss
EMBOSS esta cargado.
Puedes teclear 'emnu' para comenzar a utilizarlo en modo menu
Ahora estan cargadas en memoria las direcciones de comandos y demas datos necesarios y podemos trabajar con Emboss.
[pgrillo@harle pgrillo]$ wossname "coiled
coil"
Finds programs by keywords in their one-line documentation
SEARCH FOR 'COILED COIL'
pepcoil Predicts coiled coil regions
Otra búsqueda con wossname:
[pgrillo@harle pgrillo]$ wossname align
Finds programs by keywords in their one-line documentation
SEARCH FOR 'ALIGN'
cons Creates a consensus from multiple alignments
distmat Creates a distance matrix from multiple alignments
ealistat Statistics for multiple alignment files
ealistat Statistics for multiple alignment files
ehmmalign Align sequences with an HMM
ehmmalign Align sequences with an HMM
ehmmpfam Align single sequence with an HMM
ehmmpfam Align single sequence with an HMM
emma Multiple alignment program - interface to ClustalW program
est2genome Align EST and genomic DNA sequences
infoalign Information on a multiple sequence alignment
matcher Finds the best local alignments between two sequences
needle Needleman-Wunsch global alignment
plotcon Plots the quality of conservation of a sequence alignment
prettyplot Displays aligned sequences, with colouring and boxing
prophecy Creates matrices/profiles from multiple alignments
prophet Gapped alignment for profiles
scopalign Generate alignments for SCOP families
showalign Display a multiple sequence alignment
stretcher Finds the best global alignment between two sequences
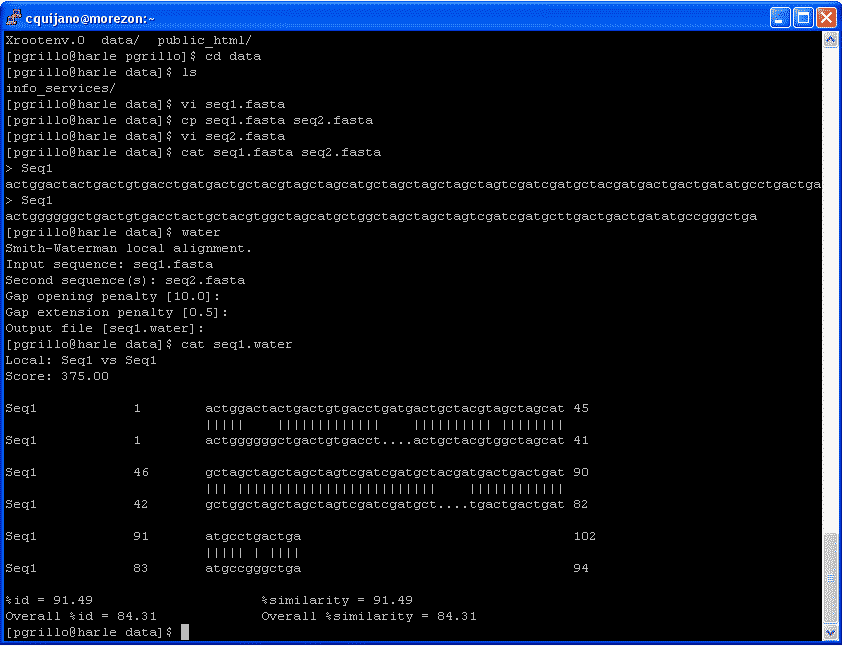
water Smith-Waterman local alignment
Podemos acceder a las aplicaciones como ejecutamos cualquier otro comando u orden del sistema:
| Servicio de BioInformatica | |
| Ultima modificación: 25 de Febrero de 2003 |